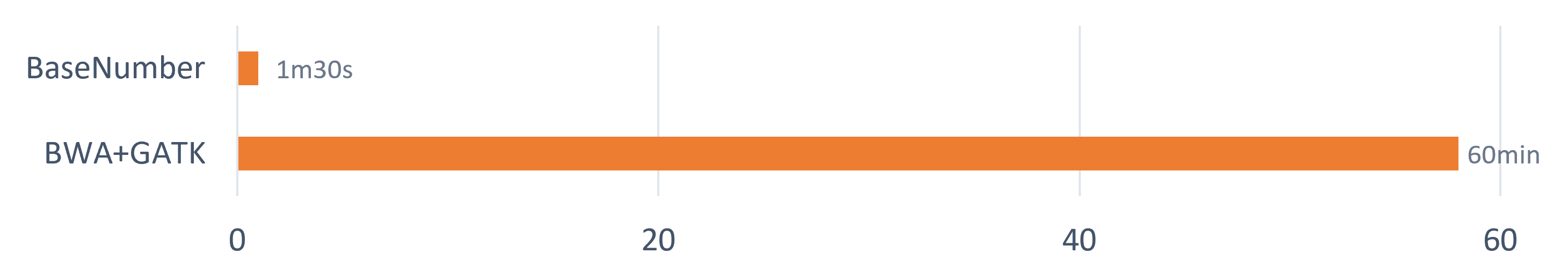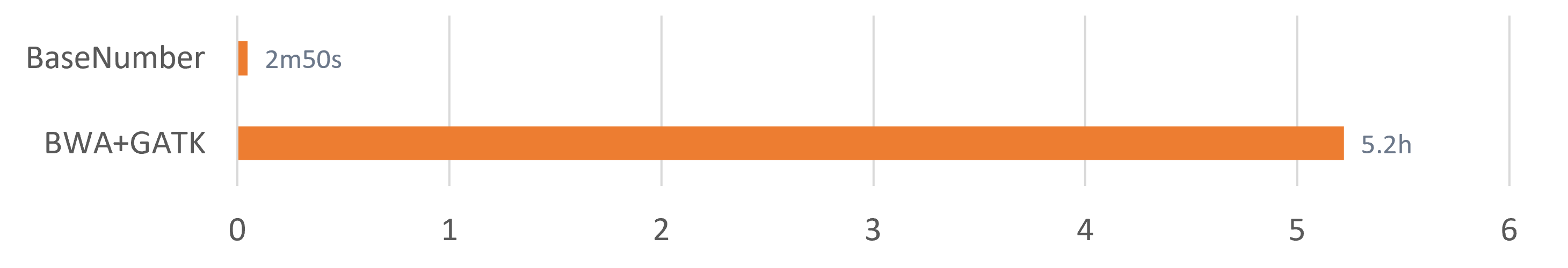BaseNumber DNA测序数据分析系统
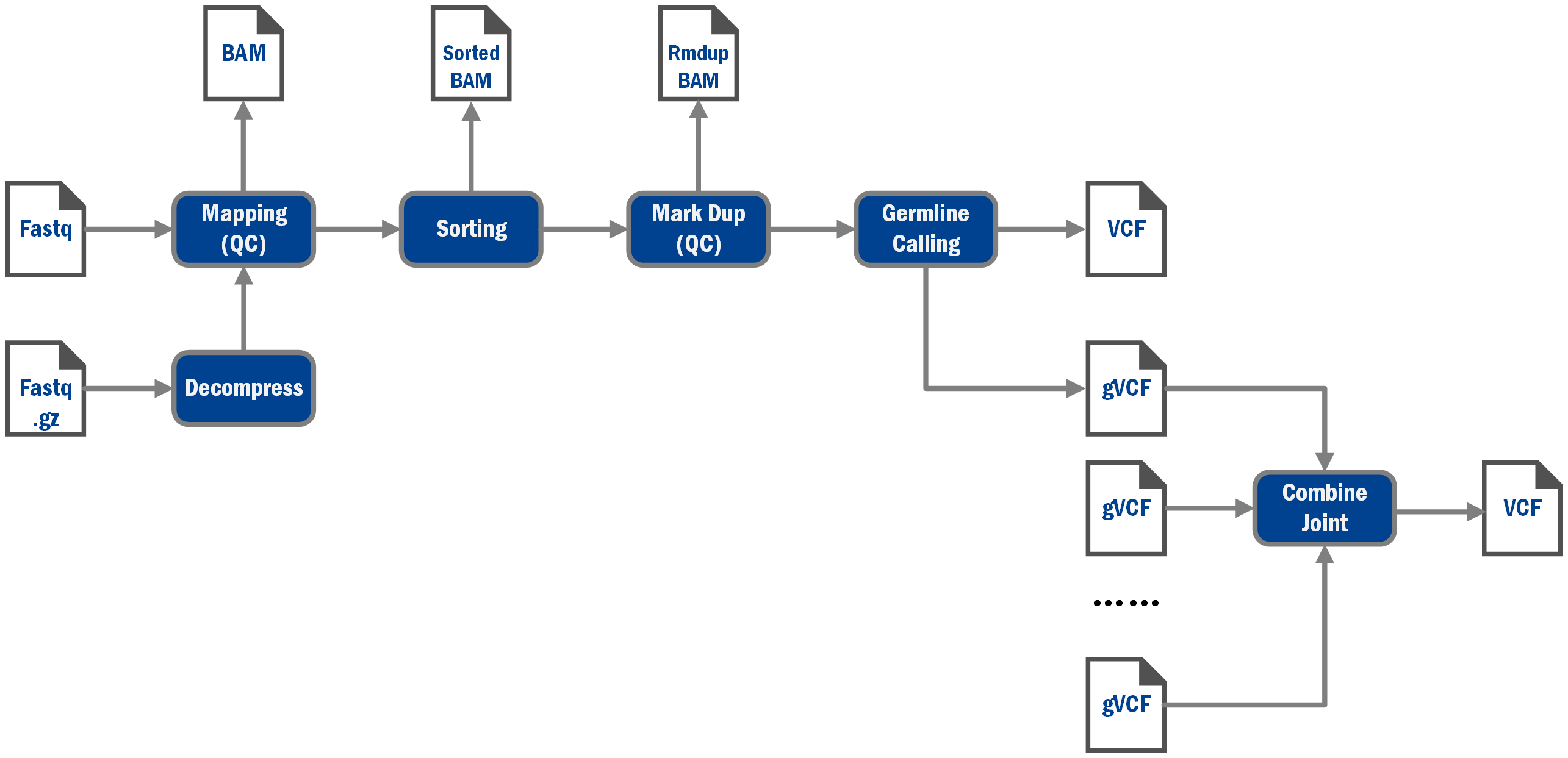
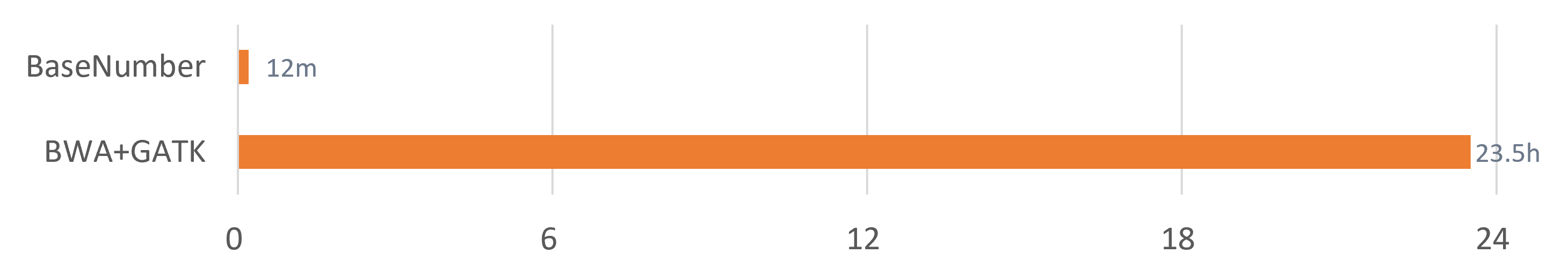
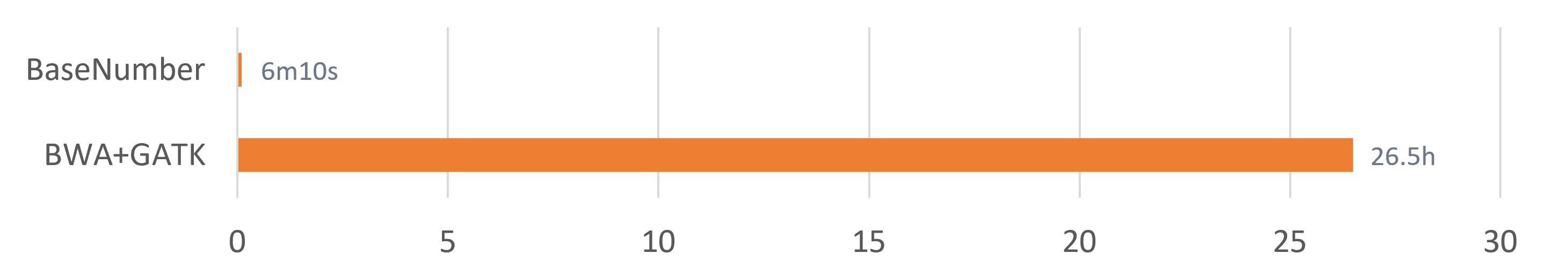
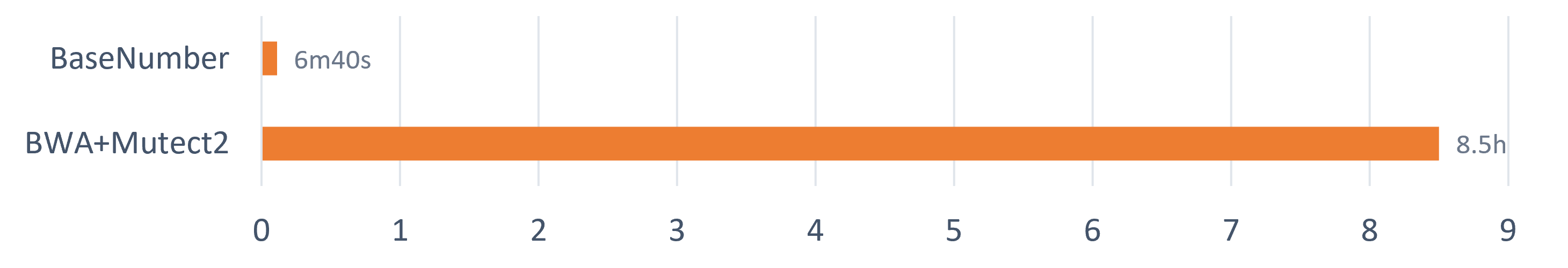
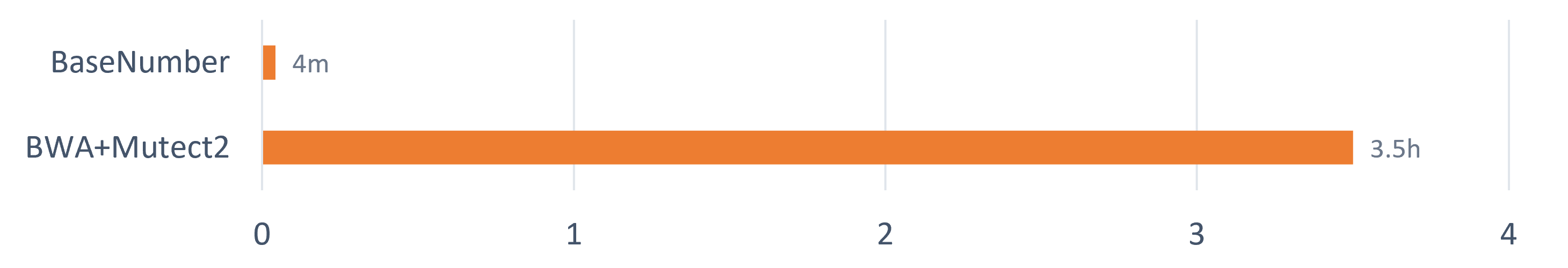
BaseNumber DNA测序数据分析系统通过执行基于“CUDA+GPU”计算环境开发的高并行算法,将DNA测序数据二级分析速度提升百倍,极大缩短计算时间,降低用户成本。
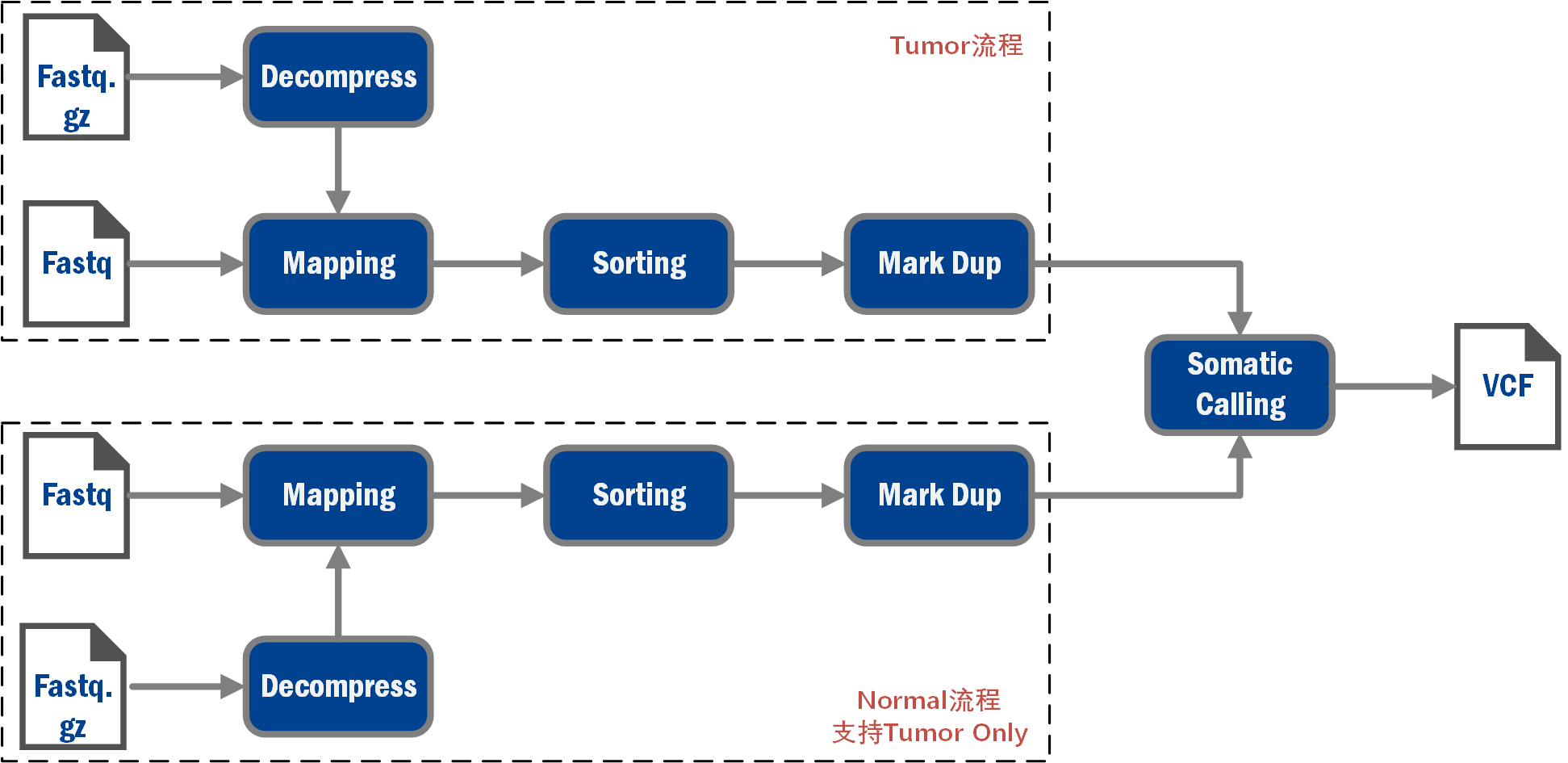
平台内置多种分析流程,助力用户全方位提升NGS数据分析能力,释放数据价值。

超高速分析

高通量、方案经济

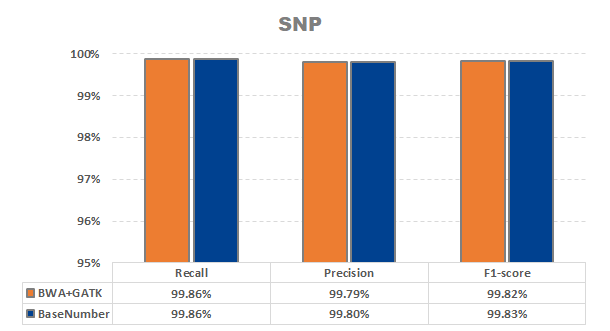
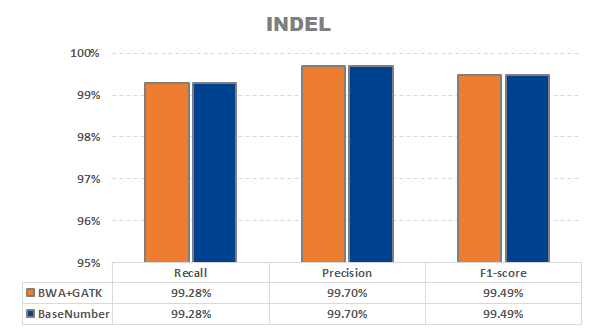
准确

易用